Un equipo de investigadores del MIT, la Universidad del Estado de Arizona, y Baylor University han desarrollado un nuevo algoritmo informático que hace el trabajo de síntesis y manipulación de ADN para hacer macromoléculas de formas geométricas deseadas . Los resultados de su investigación han sido publicados en la revista Science.
"El estudio se centra en evitar el problema de que sea necesario un experto que diseñe el ADN para sintetizar el objeto, pasando a que el objeto en sí es el punto de partida, con las secuencias de ADN que son necesarias definidas automáticamente por un algoritmo".
Resumen de la publicación:
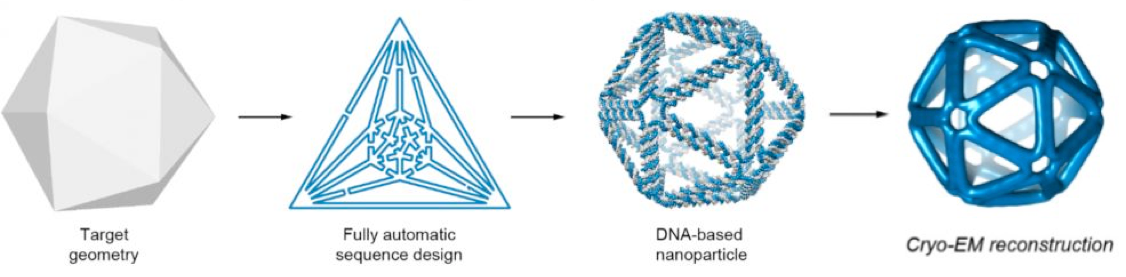
Soportes en forma de origami de ADN es un medio versátil de la síntesis de arquitecturas moleculares complejas. Sin embargo, el enfoque está limitado por la necesidad de hacer el diseño Watson-Crick de apareamiento de bases manualmente para cualquier estructura final dada.
Aquí, se presenta una estrategia general de arriba hacia abajo para diseñar arquitecturas de ADN casi arbitrarias de manera autónoma basándose sólo en forma destino. Los objetos se representan como superficies cerradas de redes poliédricas de dúplex de ADN en paralelo, lo que permite el enrutamiento completo de la estructura de ADN con un algoritmo de árbol de expansión.
Se aplicó la reacción en cadena de la polimerasa asimétrica para producir los conjuntos estables, con una longitud de estructura personalizado y la secuencia que se verifica estructuralmente en 3D para ser de alta fidelidad utilizando una sola partícula de crio-microscopía electrónica. Su estabilidad a largo plazo en el suero y tampón bajo en sal confirma su utilidad para biológica, así como las aplicaciones no biológicas.
futurism.com/scientists-find-a-better-way-to-make-structures-and-create-dna-based-tech/